Objekte anlegen in R
Objekte kann man ganz leicht anlegen:
Objektname <- FunktionDas klingt jetzt etwas abstrakt. Ein Objekt ist einfach eine Speichereinheit um das zu speichern, was die Funktion angibt. Das kann zum Beispiel eine Rechenoperation sein.
a <- 1+1 Das Objekt a enthält jetzt den Wert 2.
Man kann hier aber auch einen Datensatz speichern.
monate <- c("Januar", "Februar", "Maerz", "April", "Mai", "Juni", "Juli", "August", "September", "Oktober", "November", "Dezember")Mit der Funktion c() fügt man dem Objekt mehrere Inhalte hinzu. Diese sind intern durchnummeriert, man kann sie aufrufen, indem man die Nummer in eckige Klammern schreibt. Um den ersten Monat abzurufen, schreibe ich in R also
monate[1]und erhalte in der Ausgabekonsole:
[1] "Januar"Wie kann ich Objekte in R wieder löschen?
Mit dem Befehl rm() kann man Objekte löschen. Also zB. rm(a), wenn man das Objekt a löschen will.
Man kann auch mehrere Objekte löschen. Wenn ich die Objekte a und monate löschen will, trenne ich sie einfach mit einem Komma:
rm(a,monate)Wenn ich alle Objekte aus meinem Environment löschen will, mache ich das mit
rm(list = ls())Wichtig ist dass ihr nur Objekte löschen könnt, die ihr auch angelegt habt. Wenn ihr a schon gelöscht habt und dann noch mal löschen wollt, bekommt ihr eine Fehlermeldung.
Daten importieren und exportieren
Man kann in RStudio auf unterschiedliche Art Datensätze importieren.
Der einfachste Weg ist oben im Reiter Environment auf Import Dataset zu klicken.
Angenommen wir haben eine Txt Datei, die einen Datensatz mit Büchern enthält, die ich gelesen habe.
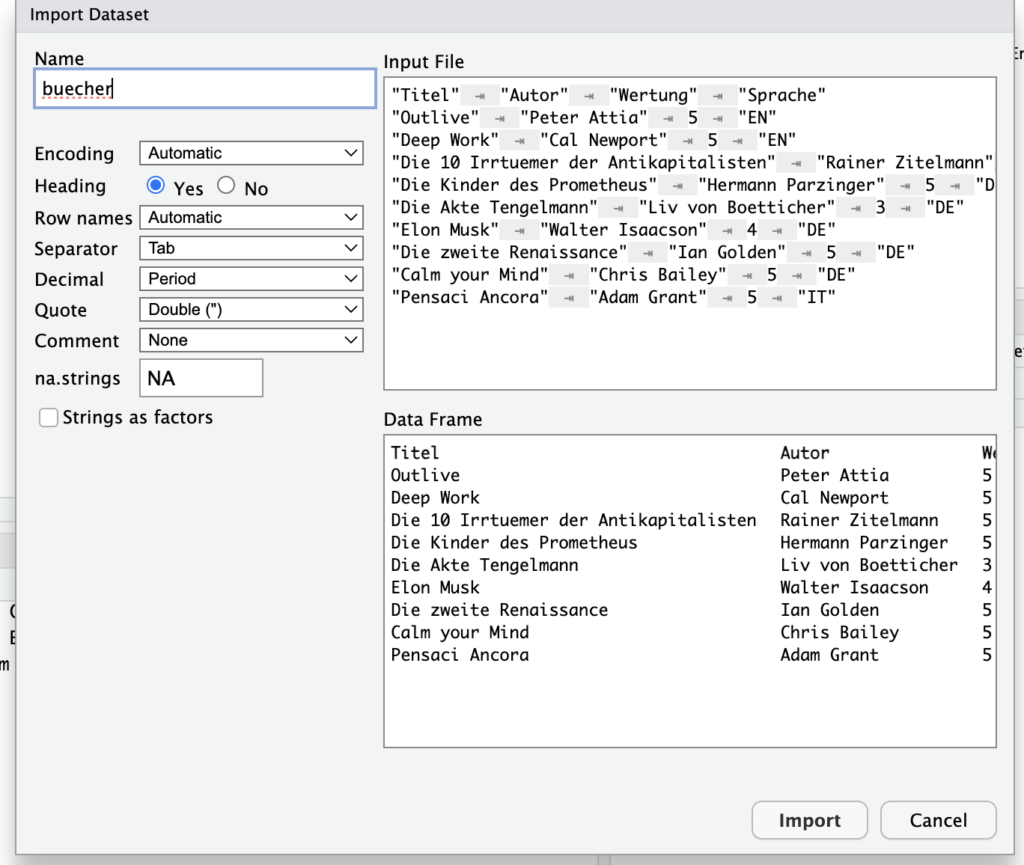
Im Idealfall erkennt RStudio eigenständig wie die einzelnen Felder getrennt sind. In meinem Fall mit dem Tab. Ansonsten könnt ihr das manuell einstellen.
Dann importiere ich das Ganze und kann jetzt auf das Objekt „buecher“ zugreifen.
Namen der Variablen in einem Objekt in R ausgeben
Ich hab jetzt natürlich schon beim Importieren gesehen, welche Variablen es gibt. Aber angenommen ich weiß es nicht (mehr) kann ich sie mir auch in R ausgeben lassen.
names(buecher)Und das Ergebnis ist:
[1] "Titel" "Autor" "Wertung" "Sprache"Wenn ich den ganzen Datensatz in einem extra Fenster anzeigen möchte, benutze ich den Befehl View() großgeschrieben.
Ich möchte nun alle Bücher, die eine Wertung von 5 haben in dem Objekt „guteBuecher“ abspeichern.
guteBuecher <- buecher[buecher$Wertung == 5,]Das Objekt guteBuecher enthält jetzt alle Einträge im Objekt Bücher, wo die Wertung 5 ist. Das Komma am Ende bedeutet, dass alle Zeilen ausgewählt werden.
Im nächsten Schritt möchte ich diese Bücher auflisten:
guteBuecher["Titel"]Das ist das Ergebnis:

Ma sieht hier, dass die Einträge 5 und 6 fehlen, weil diese Bücher weniger als 5 Sterne bekommen haben.
Mein Objekt in R in einer neuen Textdatei speichern
Jetzt möchte ich die Titel der guten Bücher in einer neuen Textdatei speichern.
Dazu verwende ich folgenden Befehl:
write.table(guteBuecher["Titel"], file = "gutebuecher.txt", sep="\t", row.names= FALSE)Normalerweise werden die einzelnen Einträge in der exportierten Datei mit einem Leerzeichen getrennt. Das möchte ich aber nicht, sondern ich möchte, dass stattdessen ein Tab verwendet wird. Das sage ich ihm, indem ich sep=“\t“ benutze.
Der Befehl row.names = False bewirkt, dass keine Zeilennummern mit exportiert werden.
So sieht meine Textdatei jetzt aus:

Da es hier nur eine einzelne Spalte gibt, hätte ich mir den Befehl sep=“\t“ natürlich sparen können 😅
Im nächsten Schritt möchte ich nur Bücher anzeigen, die ich NICHT auf deutsch gelesen habe. Dafür verwende ich statt == den Operator !=
buecher[buecher$Sprache != "DE",]Das Komma am Ende sorgt wieder dafür, dass alle Spalten angezeigt werden.
Ich kann mir aber auch einfach nur die erste Spalte ausgeben lassen, also den Titel:
buecher[buecher$Sprache != "DE",1]Das sieht dann so aus:
[1] "Outlive" "Deep Work" "Pensaci Ancora"Die Struktur eines Objekts in R ausgeben
Mit dem Befehl str() kann man sich die Struktur eines Objekts ausgeben.
Für das Objekt buecher sieht das so aus:
> str(buecher)'data.frame': 9 obs. of 4 variables: $ Titel : chr "Outlive" "Deep Work" "Die 10 Irrtuemer der Antikapitalisten" "Die Kinder des Prometheus" ... $ Autor : chr "Peter Attia" "Cal Newport" "Rainer Zitelmann" "Hermann Parzinger" ... $ Wertung: int 5 5 5 5 3 4 5 5 5 $ Sprache: chr "EN" "EN" "DE" "DE" ...Die Punkte bedeuten, dass da noch weitere Einträge folgen.
Für uns interessant ist aber erstmal interessant, in was für einer Art Variablen gespeichert wurden.
Wir sehen hier, dass Titel, Autor und Sprache die Bezeichnung chr haben. Das heißt, es sind character Variablen, sie speichern also Text. Wertung ist eine Int-Variable, also eine Zahl. Hätte ich in meiner Ursprungsdatei die Wertungen in Anführungszeichen geschrieben, wären sie auch als Character – also als Textvariablen – gespeichert worden.
Das hätte den Nachteil gehabt, dass ich mit Textvariablen nicht rechnen kann.
Ich könnte nämlich jetzt alle Wertungen miteinander addieren und durch die Anzahl der Bewertungen teilen, um einen Durchschnitt zu errechen.
Die einfachste Methode ist:
alleBewertungenZusammengerechnet <- buecher$Wertung[1] + buecher$Wertung[2] + buecher$Wertung[3] + buecher$Wertung[4] + buecher$Wertung[5] + buecher$Wertung[6] + buecher$Wertung[7] + buecher$Wertung[8] + buecher$Wertung[9]
alleBewertungenZusammengerechnet / 9Hier erzeuge ich ein Objekt das alleBewertungenZusammengerechnet heißt und die Bewertungen enthält. Ich addiere einfach den Wert aus „Bewertung“ der einzelnen Zeilen.
Wohin speichert R die Dateien?
Wenn ich eine neue Datei erzeuge oder ein Objekt als Datei abspeicher, speichert R die Datei im Arbeitsverzeichnis.
Um herauszufinden, wo das Arbeitsverzeichnis ist (falls ich das nicht weiß, was ich aber meistens weiß) kann ich den Befehl
getwd()verwenden.
Wenn ich die Datei in ein anderes Arbeitsverzeichnis speichern will, mache ich das mit:
setwd()Also zum Beispiel: setwd(„/Users/deinname/Downloads“)
hierbei die Anführungszeichen beachten!
Wenn ihr dann eine Datei abspeichert, landet sie in diesem Ordner.
Pakete installieren und Laden in R
Standardmäßig sind schon einige Pakete in R aktiviert. In R Studio kann man unter Packages sehen, welche Pakete installiert sind. Weitere Pakete kann man mit dem Befehl:
install.packages()installieren. Dabei schreibt man den Paketnamen in Anführungszeichen.
Zum Beispiel so, um das Car-Paket zu laden:
install.packages("car")Anschließend muss man das Paket noch laden, das geht so:
library()
Diesmal ohne Anführungszeichen.
In RStudio kann man Pakete auch über einen Klick auf „Install“ installieren.
Wie kann ich mehrere Pakete in RStudio gleichzeitig laden?
Mit dem Befehl lapply. Man verbindet die Namen der Pakete mit dem c Befehl
lapply(c("Paketname1", "Paketname2", "Paketname3"), library, character.only = TRUE)Wie rufe ich in R die Hilfe auf?
Man kann die Hilfe zu einem Befehl mit einem Fragezeichen aufrufen. Zum Beispiel so:
?strDann erhält man die Hilfe zum Befehl str().
Man kann aber auch nach Stichwörtern in der Hilfe suchen. Diesmal mit 2 Fragezeichen.
??structureSo suche ich nach der Hilfe zum Stichwort structure.

Schreibe einen Kommentar